5.2 Eksperimen Algoritma K-Medoids
Data
Dengan menggunakan fungsi read.csv() dari R, kami dapat mengimpor data langsung dari URL tersebut ke dalam lingkungan kerja R. Berikut adalah kode yang digunakan untuk membaca data: Package reader menyiapkan fungsi read_csv() untuk import data dari file CSV. Pada kasus ini digunakan data Data 40% Kemiskinan di jawa Tengah.
library (readr)
urlfile = "https://bit.ly/3VO3kRE"
data<-read.csv(url(urlfile), row.names = "Kabupaten")| X1 | X2 | X3 | X4 | X5 | X6 | X7 | X8 | X9 | X10 | |
|---|---|---|---|---|---|---|---|---|---|---|
| CILACAP | 5.19 | 5.67 | 5.08 | 5.44 | 5.22 | 6.05 | 11.47 | 9.78 | 5.55 | 5.12 |
| BANYUMAS | 5.71 | 4.47 | 5.18 | 5.51 | 5.02 | 6.21 | 7.39 | 6.96 | 5.98 | 8.22 |
| PURBALINGGA | 3.30 | 2.19 | 3.80 | 3.13 | 3.73 | 3.34 | 8.71 | 7.41 | 3.21 | 4.65 |
| BANJARNEGARA | 2.73 | 2.34 | 3.76 | 2.80 | 2.57 | 2.99 | 3.31 | 5.45 | 4.21 | 6.05 |
| KEBUMEN | 4.17 | 2.55 | 3.26 | 4.16 | 3.15 | 4.15 | 4.30 | 9.29 | 4.61 | 4.34 |
| PURWOREJO | 1.87 | 2.12 | 1.48 | 3.05 | 1.78 | 1.83 | 5.00 | 4.90 | 3.12 | 2.09 |
| WONOSOBO | 2.13 | 1.95 | 3.00 | 1.78 | 1.62 | 2.06 | 0.45 | 2.32 | 3.57 | 0.84 |
| MAGELANG | 3.95 | 3.01 | 4.22 | 4.15 | 3.01 | 3.64 | 1.44 | 3.35 | 5.69 | 3.67 |
| BOYOLALI | 2.19 | 3.07 | 1.61 | 2.74 | 2.11 | 1.82 | 1.71 | 2.34 | 3.41 | 1.55 |
| KLATEN | 3.84 | 5.15 | 1.93 | 4.64 | 4.04 | 3.78 | 8.71 | 4.45 | 3.99 | 3.09 |
R packages dan fungsi yang dibutuhkan
Untuk melakukan analisis cluster dengan metode k-medoids di R, diperlukan beberapa paket dan fungsi utama. Paket yang wajib digunakan adalah cluster, yang menyediakan fungsi pam() (Partitioning Around Medoids) sebagai implementasi utama algoritma k-medoids. Selain itu, paket factoextra berguna untuk visualisasi hasil clustering dengan fungsi fviz_cluster(). Untuk mengevaluasi kualitas clustering, paket fpc dan clusterCrit dapat digunakan sebagai opsi tambahan dengan fungsi seperti cluster.stats() untuk evaluasi statistik dan intCriteria() untuk menghitung indeks validasi clustering. Dalam proses analisis, fungsi silhouette() dapat digunakan untuk mengevaluasi kualitas clustering dengan Silhouette Width, sementara dist() diperlukan untuk membuat matriks jarak yang digunakan oleh algoritma. Sebagai langkah awal, data biasanya diskalakan menggunakan fungsi seperti scale() sebelum diterapkan algoritma k-medoids untuk memastikan hasil yang optimal. Kombinasi paket dan fungsi ini memungkinkan analisis cluster yang efektif, mulai dari pembentukan cluster hingga evaluasi hasil.
Estimasi Jumlah Cluster Optimal
Untuk menentukan jumlah cluster yang optimal, metode average silhouette dapat digunakan. Konsepnya adalah menjalankan algoritma PAM dengan berbagai jumlah cluster \(k\). Kemudian, rata-rata silhouette dari masing-masing cluster dihitung dan diplot berdasarkan jumlah cluster. Average silhouette digunakan untuk menilai kualitas clustering, di mana nilai yang tinggi menunjukkan clustering yang baik. Jumlah cluster \(k\) yang optimal adalah yang menghasilkan rata-rata silhouette tertinggi dalam rentang nilai \(k\) yang dipertimbangkan.
Fungsi fviz_nbclust() dari paket factoextra menyediakan cara praktis untuk memperkirakan jumlah cluster yang optimal. Dengan memanfaatkan metode average silhouette, fungsi ini dapat membantu menentukan jumlah cluster yang menghasilkan hasil clustering terbaik. Contoh implementasinya adalah sebagai berikut:
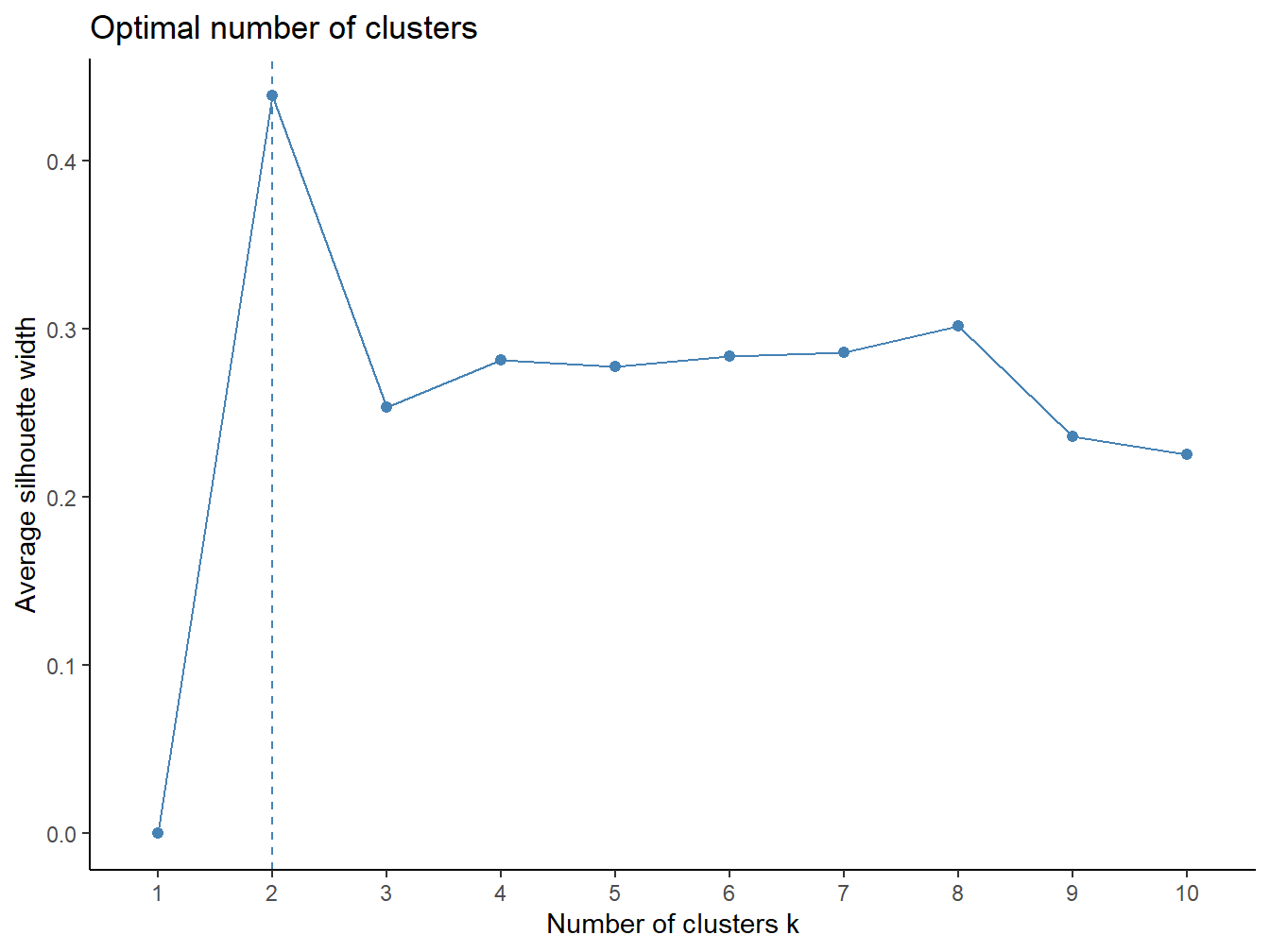
Gambar 5.1: Plot Jumlah Cluster Metode silhouette
Hasilnya adalah grafik yang menampilkan nilai rata-rata silhouette untuk setiap jumlah cluster. Jumlah cluster optimal ditentukan oleh nilai silhouette tertinggi, yang menunjukkan pembagian cluster terbaik dalam data.Berdasarkan plot, jumlah cluster yang disarankan adalah 2. Pada langkah berikutnya, kita akan mengelompokkan observasi ke dalam 2 cluster.
Eksperimen K-Medoids Clustering
Berdasarkan plot hasil analisis dengan metode average silhouette, jumlah cluster optimal yang disarankan adalah 2. Oleh karena itu, dalam analisis cluster menggunakan algoritma k-medoids, kita akan menggunakan jumlah cluster \(k = 2\). Langkah ini diharapkan dapat menghasilkan pembagian cluster yang optimal dan berkualitas tinggi sesuai dengan data yang dianalisis. Berikut adalah kode untuk menjalankan algoritma k-medoids dengan \(k = 2\) menggunakan fungsi pam() dan mencetak hasilnya:
Kode ini akan menampilkan detail hasil clustering, termasuk informasi tentang medoid, anggota cluster, dan ukuran setiap cluster.
# Mencetak hasil clustering
print(pam.res)
#> Medoids:
#> ID X1 X2 X3 X4 X5 X6 X7 X8 X9 X10
#> TEGAL 28 4.78 3.91 6.70 4.84 6.35 5.82 3.00 5.99 2.98 5.98
#> SEMARANG 22 1.76 2.19 1.61 2.33 1.88 1.61 0.92 0.49 2.67 1.56
#> Clustering vector:
#> CILACAP BANYUMAS PURBALINGGA BANJARNEGARA KEBUMEN
#> 1 1 1 1 1
#> PURWOREJO WONOSOBO MAGELANG BOYOLALI KLATEN
#> 2 2 2 2 1
#> SUKOHARJO WONOGIRI KARANGANYAR SRAGEN GROBOGAN
#> 2 2 2 2 1
#> BLORA REMBANG PATI KUDUS JEPARA
#> 2 2 2 2 1
#> DEMAK SEMARANG TEMANGGUNG KENDAL BATANG
#> 2 2 2 2 2
#> PEKALONGAN PEMALANG TEGAL BREBES KOTA MAGELANG
#> 2 1 1 1 2
#> KOTA SURAKARTA KOTA SALATIGA KOTA SEMARANG KOTA PEKALONGAN KOTA TEGAL
#> 2 2 2 2 2
#> Objective function:
#> build swap
#> 4.702218 4.605625
#>
#> Available components:
#> [1] "medoids" "id.med" "clustering" "objective" "isolation"
#> [6] "clusinfo" "silinfo" "diss" "call" "data"Hasil analisis k-medoids dengan \(k = 2\) menunjukkan bahwa data berhasil dikelompokkan ke dalam dua cluster. Medoid yang terpilih sebagai representasi masing-masing cluster adalah TEGAL untuk Cluster 1 dan SEMARANG untuk Cluster 2, dengan nilai atribut masing-masing ditampilkan dalam tabel. Observasi lainnya dikelompokkan berdasarkan kedekatan mereka dengan medoid, misalnya CILACAP, BANYUMAS, dan PURBALINGGA masuk ke Cluster 1, sementara PURWOREJO, WONOSOBO, dan MAGELANG masuk ke Cluster 2. Secara keseluruhan, clustering ini memberikan pembagian yang optimal dengan dua cluster yang masing-masing diwakili oleh medoid terpilih.
Untuk menambahkan klasifikasi titik (cluster) ke data asli, Anda dapat menggunakan kode berikut. Kode ini akan menggabungkan hasil klasifikasi (cluster) dari analisis k-medoids ke dalam data dan menampilkan 3 baris pertama dari data yang telah diperbarui:
# Menambahkan klasifikasi cluster ke data asli
hasil_cluster <- cbind(data, cluster = pam.res$cluster)
# Menampilkan 3 baris pertama
head(hasil_cluster, n = 3)
#> X1 X2 X3 X4 X5 X6 X7 X8 X9 X10 cluster
#> CILACAP 5.19 5.67 5.08 5.44 5.22 6.05 11.47 9.78 5.55 5.12 1
#> BANYUMAS 5.71 4.47 5.18 5.51 5.02 6.21 7.39 6.96 5.98 8.22 1
#> PURBALINGGA 3.30 2.19 3.80 3.13 3.73 3.34 8.71 7.41 3.21 4.65 1Hasil dari kode ini akan menampilkan tiga baris pertama dari data, dengan kolom tambahan yang menunjukkan cluster tempat setiap observasi dikelompokkan. Kolom baru cluster berisi angka 1 atau 2 yang mewakili dua cluster yang terbentuk.
Visualisasi K-Medoids Clustering
Untuk memvisualisasikan hasil pengelompokan dengan k-medoids, kita akan menggunakan fungsi fviz_cluster() dari paket factoextra. Fungsi ini akan menghasilkan plot sebar titik data yang diwarnai sesuai dengan nomor cluster. Dalam kode ini, palet warna yang digunakan adalah #00AFBB untuk cluster pertama dan #FC4E07 untuk cluster kedua. Elips konsentrasi ditambahkan dengan parameter ellipse.type = “t”, yang menunjukkan distribusi data di setiap cluster. Selain itu, untuk menghindari tumpang tindih label, parameter repel = TRUE digunakan, meskipun ini dapat sedikit memperlambat proses visualisasi. Terakhir, tema plot yang klasik diterapkan dengan ggtheme = theme_classic(), memberikan tampilan yang bersih dan sederhana pada grafik.
fviz_cluster(pam.res,
palette = c("#00AFBB", "#FC4E07"), # Palet warna
ellipse.type = "t", # Elips konsentrasi
repel = TRUE, # Menghindari label bertumpuk (proses ini dapat sedikit lebih lambat)
ggtheme = theme_classic() # Tema plot klasik
)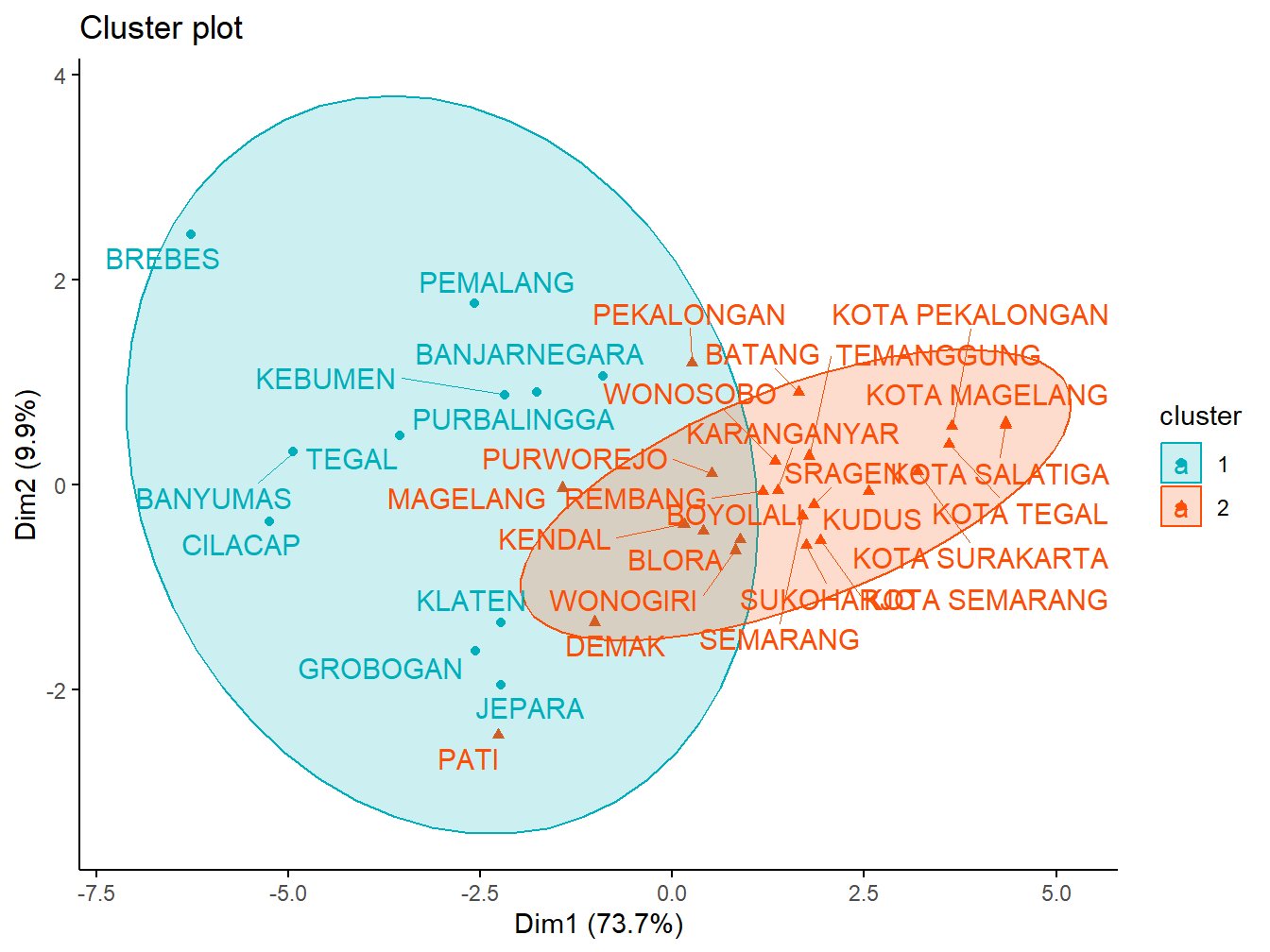
Gambar 5.2: Visualisasi Cluster K-Medoids